一提到卷积,相信大部分人和我一样,首先想到的是时下非常火的一个机器学习算法——卷积神经网络(CNN)。实际上,即使自己之前看过相关的介绍,但是对于卷积操作一直是一知半解的状态,在充分查阅相关的资料后,打算整理成一篇博客,详细介绍卷积操作及其应用。
最早接触卷积操作,应该是在学习微积分时,教材中直接抛出卷积的定义:
对于两个函数 f(t) 和 g(t),它们之间的卷积操作定义为:
f ( t ) ∗ g ( t ) = ∫ − ∞ ∞ f ( τ ) g ( t − τ ) d τ f(t)\ast g(t) = \int_{-\infty}^{\infty}f(\tau)g(t-\tau)d\tau
f ( t ) ∗ g ( t ) = ∫ − ∞ ∞ f ( τ ) g ( t − τ ) d τ
尽管这个定义比较清晰,但是我们很难从中发掘出其背后的实际意义。下面让我们从概率论的角度挖掘卷积操作背后的内涵。
设 X,Y 为两个独立变量,如果我们要计算 Z=X+Y 的概率, 可以这样理解,对于每一个确定的 z,存在无数组 x、y 满足条件使得 x+y=z,因此我们需要将这些概率累积求和,如果 X 和 Y 是离散的变量,其概率分别为 p x ( x ) p_x(x) p x ( x ) p y ( y ) p_y(y) p y ( y )
p z ( z ) = ∑ x = − ∞ ∞ p x ( x ) p y ( z − x ) p_z(z) = \sum^{\infty}_{x=-\infty}p_x(x)p_y(z-x)
p z ( z ) = x = − ∞ ∑ ∞ p x ( x ) p y ( z − x )
对于 X 和 Y 是连续的情况,其概率密度函数分别是 f x ( x ) f_x(x) f x ( x ) f y ( y ) f_y(y) f y ( y )
f z ( z ) = ∫ − ∞ ∞ f x ( x ) f y ( z − x ) d x f_z(z) = \int^{\infty}_{-\infty}f_x(x)f_y(z-x)dx
f z ( z ) = ∫ − ∞ ∞ f x ( x ) f y ( z − x ) d x
可以看到,以上两个公式正好是卷积的离散形式和连续形式的定义。因此,卷积操作与概率和统计中求独立随机变量的和完全等价 。更加生动形象的例子可以参看丢骰子 的例子,这里不再赘述。
numpy 和 scipy 中都提供了卷积计算的相关函数定义,不同的是,scipy 中除了直接求和进行卷积计算之外,还提供了通过快速傅里叶变换的方式来计算卷积,这种方法对于较大尺寸的数组之间的卷积计算速度更快,其原理是卷积定理 。下面将主要介绍 numpy 中卷积计算。
numpy.convolve 函数有三个输入参数,a,v 和 mode,其中 a 和 v 是输入矩阵,需要注意的一点是,官方文档中特别指出如果数组 v 的长度大于数组 a,那么在计算之前会交换两个数组,换句话讲,在计算的过程中始终保持数组 a 的长度大于数组 v。
full 模式full 模式会计算数组 a 和 v 的所有卷积,其结果包含了所有可能重叠的部分,结果长度为:
l e n g t h = l e n ( a ) + l e n ( v ) − 1 length = len(a) + len(v) -1
l e n g t h = l e n ( a ) + l e n ( v ) − 1
示例代码和输出为:
1 2 3 4 5 import numpy as npa = np.array([1 , 2 , 3 , 4 , 5 ]) v = np.array([0.0 , 0.5 , 1.0 ]) np.convolve(a, v, mode='full' )
array([0. , 0.5, 2. , 3.5, 5. , 6.5, 5. ])
在输出数组的前两个和最后两个元素并不是a和v的所有元素卷积和,而只包含部分重叠部分求和。
same 模式same 模式会返回与输入数组 a 长度相同的卷积结果,边界部分会被截断,即是:
l e n g t h = l e n ( a ) length = len(a)
l e n g t h = l e n ( a )
示例代码和输出:
1 2 3 4 5 import numpy as npa = np.array([1 , 2 , 3 , 4 , 5 ]) v = np.array([0.0 , 0.5 , 1.0 ]) np.convolve(a, v, mode='same' )
array([0.5, 2. , 3.5, 5. , 6.5])
valid 模式valid 模式只会计算完全重叠部分的卷积,会直接舍弃不完全重叠的结果,其结果长度为:
l e n g t h = l e n ( a ) − l e n ( v ) + 1 length = len(a) - len(v) + 1
l e n g t h = l e n ( a ) − l e n ( v ) + 1
示例代码和输出为:
1 2 3 4 5 import numpy as npa = np.array([1 , 2 , 3 , 4 , 5 ]) v = np.array([0.0 , 0.5 , 1.0 ]) np.convolve(a, v, mode='valid' )
array([2. , 3.5, 5. ])
关于 numpy.convolve 函数的计算逻辑可以参看这篇博客:https://www.cnblogs.com/redtank/p/15678714.html 。
滑动窗口算法的原理是通过维护一个特定长度的窗口,使其在整个数组上移动,每次我们只操作窗口内部的数据,通过对窗口内数据采取特定的加权平均方式,可以实现滑动窗口滤波的效果。
卷积计算本质上正是一个滑动窗口的计算模型,在计算中,a 是整个数据,而 v 是滑动窗口,下面基于卷积计算来实现一个简单的滤波平均器。
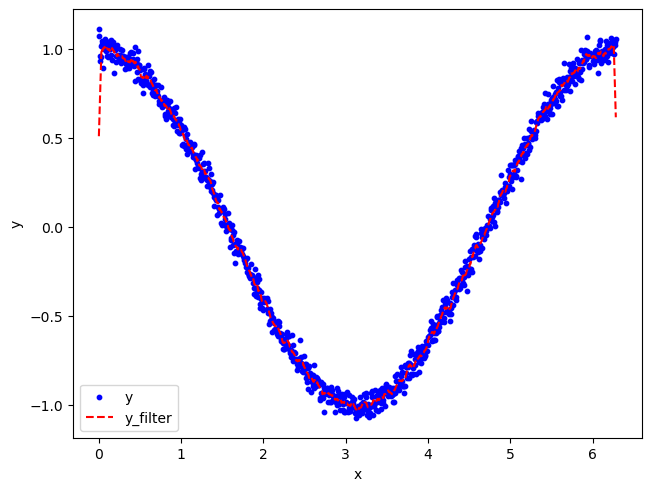
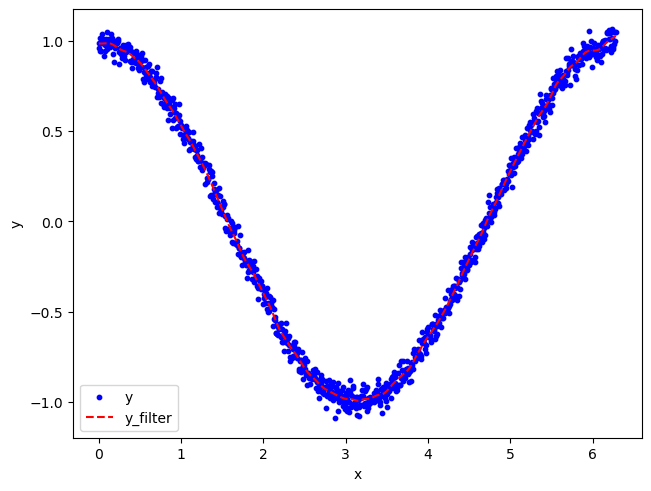
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 import numpy as npimport matplotlib.pyplot as pltx = np.linspace(0 , np.pi*2.0 , 1000 ) y = np.cos(x) + np.random.normal(0 , 0.2 , x.shape)*0.2 v = np.ones(10 ) v = v/np.sum (v) y_filter = np.convolve(y, v, mode='same' ) fig, axs = plt.subplots(dpi=100 , layout='constrained' ) axs.scatter(x, y, marker='o' , s=10 , color='blue' , label='y' ) axs.plot(x, y_filter, color='red' , linewidth=1.5 , linestyle='dashed' , label='y_filter' ) axs.set_xlabel('x' ) axs.set_ylabel('y' ) axs.legend()
运行结果:
可以看到,经过滤波处理之后曲线的平滑度得到大大改善,基本能代表真实信号,在整个过程中,v 相当于是一个在输入信号 y 中滑动的窗口,对窗口内的数据进行加权平均操作产生过滤之后的信号,值得注意的是由于边缘效应 ,曲线的边缘部分并不能反映真实的情况,要解决这个问题,就需要对初始输入数据进行填充(padding)操作。
具体代码为:
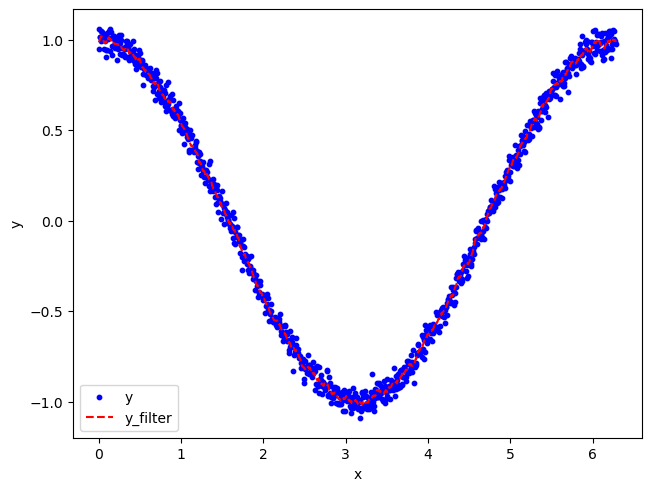
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 import numpy as npimport matplotlib.pyplot as pltx = np.linspace(0 , np.pi*2.0 , 1000 ) y = np.cos(x) + np.random.normal(0 , 0.2 , x.shape)*0.2 v = np.ones(10 ) v = v/np.sum (v) pad_width = (int ((len (v)-1 )/2.0 ), len (v)-1 -int ((len (v)-1 )/2.0 )) y_pad = np.pad(y, pad_width, mode='edge' ) y_filter = np.convolve(y_pad, v, mode='valid' ) fig, axs = plt.subplots(dpi=100 , layout='constrained' ) axs.scatter(x, y, marker='o' , s=10 , color='blue' , label='y' ) axs.plot(x, y_filter, color='red' , linewidth=1.5 , linestyle='dashed' , label='y_filter' ) axs.set_xlabel('x' ) axs.set_ylabel('y' ) axs.legend()
相较于之前的代码,上面代码中对输入数据 y 首先进行了 pad 操作,使用边缘值进行填充,并且需要保证填充之后的数据y_pad与v进行卷积之后得到的结果长度等于初始输入数据 。以下为运行结果:
可以看到在使用填充后的数据与 v 进行卷积操作之后,能够较好地消除边缘效应 的影响,曲线的边缘部分基本也能够代表真实情况。
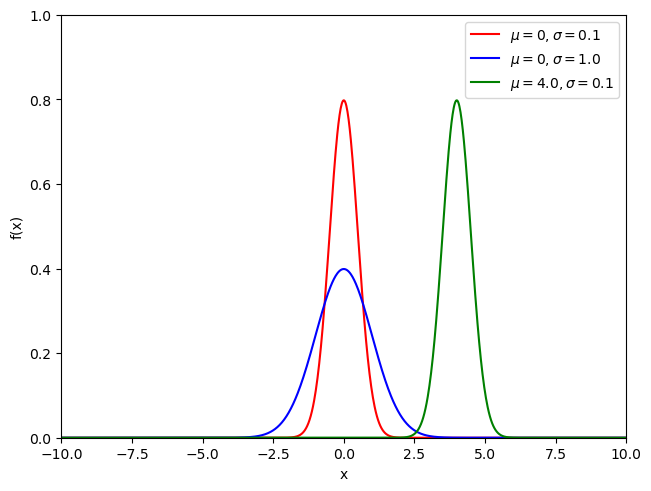
在上面的计算中,我们只是使用了最简单的取平均的方式来进行滤波,聪明的读者可能已经想到,如果我们更换窗口中数据加权平均的方式,不是就能构造出新的滤波器?高斯滤波就是这样产生的。提到高斯分布就必须要介绍一下高斯分布(正态分布),以往的实践和研究都表明,自然界中的大部分统计现象,都服从高斯分布,具体解释可以参看这篇博文:https://ruanyifeng.com/blog/2017/08/normal-distribution.html 。对于一维高斯分布,其概率密度函数的形式为:
f ( x ) = 1 σ 2 π e − ( x − μ ) 2 2 σ 2 f(x) = \frac{1}{\sigma \sqrt{2\pi}}e^{-\frac{(x-\mu)^2}{2\sigma ^2}}
f ( x ) = σ 2 π 1 e − 2 σ 2 ( x − μ ) 2
其中μ \mu μ σ \sigma σ x = μ x=\mu x = μ
其中μ \mu μ σ \sigma σ https://docs.scipy.org/doc/scipy/reference/generated/scipy.stats.multivariate_normal.html#scipy.stats.multivariate_normal ,
f ( x ) = 1 ( 2 π ) k d e t ∑ e − 1 2 ( x − μ ) T ∑ − 1 ( x − μ ) f(x) = \frac{1}{\sqrt{(2\pi)^kdet\sum}}e^{-\frac{1}{2}(x-\mu)^T\sum^{-1}(x-\mu)}
f ( x ) = ( 2 π ) k d e t ∑ 1 e − 2 1 ( x − μ ) T ∑ − 1 ( x − μ )
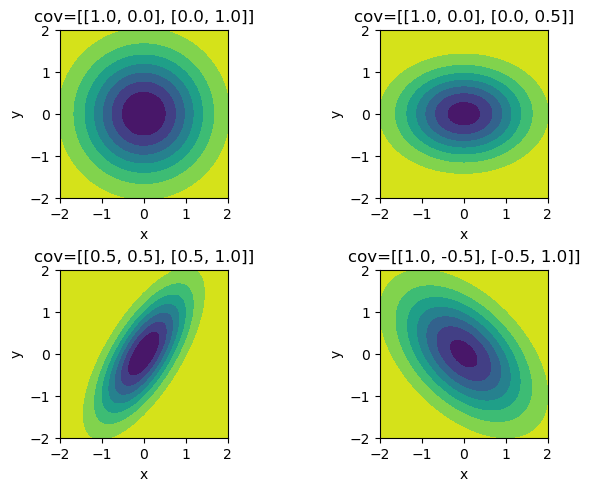
其中μ \mu μ ∑ \sum ∑ k k k ∑ \sum ∑
从上面可以总结出来,对于二维的情况,协方差矩阵的对角值决定椭圆ab轴的长度,而非对角值决定椭圆的倾斜度,这里需要注意的是要求协方差矩阵的半正定阵,对于二维的情况,需要满足:
C 11 > 0 & C 11 C 22 − C 12 C 21 > 0 C_{11} > 0 \quad\&\quad C_{11}C_{22}-C_{12}C{21}>0
C 1 1 > 0 & C 1 1 C 2 2 − C 1 2 C 2 1 > 0
言归正传,对于高斯滤波,核心思想是修改卷积过程中输入变量 v,使得从求普通平均变成求高斯平均,相关代码为:
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 import numpy as npimport matplotlib.pyplot as pltfrom scipy.stats import normx = np.linspace(0 , np.pi*2.0 , 1000 ) y = np.cos(x) + np.random.normal(0 , 0.2 , x.shape)*0.2 sigma = 1.0 v_size = int (6 *sigma+1 ) mu = v_size//2 v_tmp = np.arange(v_size) v= norm.pdf(v_tmp, loc=mu, scale=sigma) v = v/np.sum (v) pad_width = (int ((len (v)-1 )/2.0 ), len (v)-1 -int ((len (v)-1 )/2.0 )) y_pad = np.pad(y, pad_width, mode='edge' ) y_filter = np.convolve(y_pad, v, mode='valid' ) fig, axs = plt.subplots(dpi=100 , layout='constrained' ) axs.scatter(x, y, marker='o' , s=10 , color='blue' , label='y' ) axs.plot(x, y_filter, color='red' , linewidth=1.5 , linestyle='dashed' , label='y_filter' ) axs.set_xlabel('x' ) axs.set_ylabel('y' ) axs.legend()
结果为:
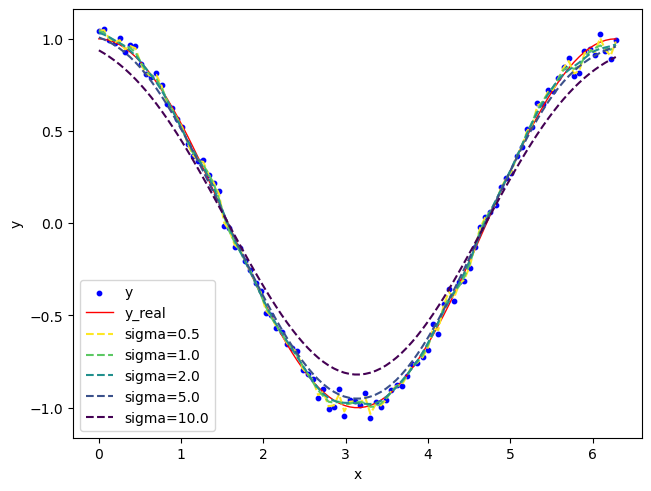
由于实践过程中误差的分布通常服从高斯分布,因此使用高斯滤波的效果通常是优于简单的平均滤波方式,下面是测试不同sigma大小对过滤结果的影响:
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 import numpy as npimport matplotlib.pyplot as pltimport matplotlib as mplfrom scipy.stats import normx = np.linspace(0 , np.pi*2.0 , 100 ) y = np.cos(x) + np.random.normal(0 , 0.2 , x.shape)*0.2 def get_v (sigma ): v_size = int (6 *sigma+1 ) mu = v_size//2 v_tmp = np.arange(v_size) v= norm.pdf(v_tmp, loc=mu, scale=sigma) v = v/np.sum (v) return v def get_gaussian_filter (y, v ): pad_width = (int ((len (v)-1 )/2.0 ), len (v)-1 -int ((len (v)-1 )/2.0 )) y_pad = np.pad(y, pad_width, mode='edge' ) y_filter = np.convolve(y_pad, v, mode='valid' ) return y_filter ls_sigma = [0.5 , 1.0 , 2.0 , 5.0 , 10.0 ] ls_v = [get_v(sigma) for sigma in ls_sigma] ls_y_filter = [get_gaussian_filter(y, v) for v in ls_v] n_lines = len (ls_sigma) cmap = mpl.colormaps['viridis_r' ] ls_colors = cmap(np.linspace(0 , 1 , n_lines)) fig, axs = plt.subplots(dpi=100 , layout='constrained' ) axs.scatter(x, y, marker='o' , s=10 , color='blue' , label='y' ) axs.plot(x, np.cos(x), color='red' , linewidth=1.0 , label='y_real' ) for i in range (len (ls_sigma)): axs.plot(x, ls_y_filter[i], color=ls_colors[i], linewidth=1.5 , linestyle='dashed' , label='sigma={}' .format (ls_sigma[i])) axs.set_xlabel('x' ) axs.set_ylabel('y' ) axs.legend()
运行结果为:
可以看到,通过调整sigma的大小,可以控制结果的平滑程度。
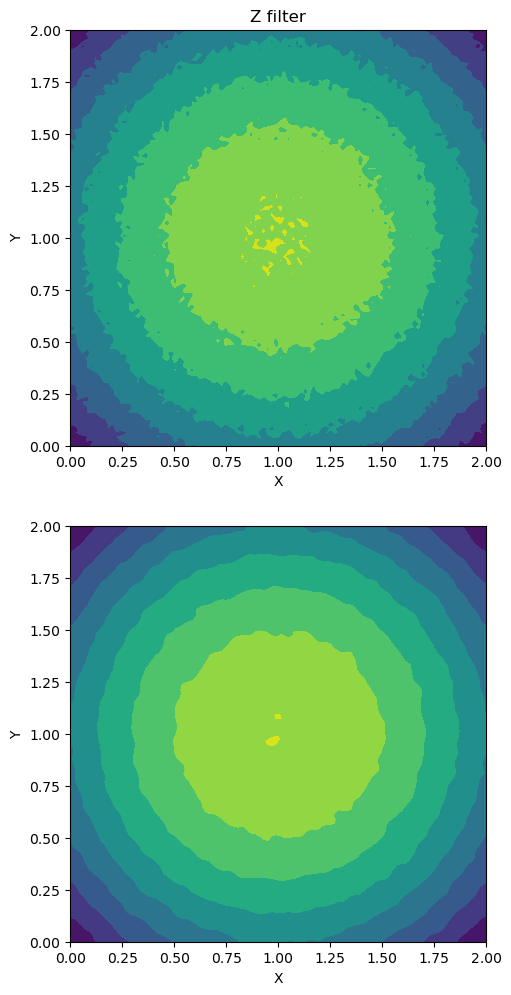
理解了以上一维的情况之后,很容易将算法扩展到更高维度,下面将以二维的情况为例。这里需要注意的一点是,numpy 中的 convolve 函数仅仅支持一维的卷积计算,要计算多维的情况,需要使用 scipy 中 提供的 convolve 方法。下面给出代码:
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 import numpy as npimport matplotlib.pyplot as pltimport matplotlib as mplfrom scipy.stats import multivariate_normalfrom scipy.signal import convolvex = np.linspace(0 , 2.0 , 100 ) y = np.linspace(0 , 2.0 , 100 ) X, Y = np.meshgrid(x, y) Z = np.power(X-1.0 , 2 ) + np.power(Y-1.0 , 2 ) + 0.1 *np.random.normal(scale=0.4 , size=X.shape) def get_v_2d (sigma1, sigma2 ): v_size1 = int (6 *sigma1+1 ) v_size2 = int (6 *sigma2+1 ) mu1 = v_size1//2 mu2 = v_size2//2 v = np.zeros((v_size1, v_size2)) for i in range (v_size1): for j in range (v_size2): v[i, j] = multivariate_normal.pdf([i, j], mean=[mu1, mu2], cov=[[sigma1, 0.0 ], [0.0 , sigma2]]) v = v/np.sum (v) return v def get_gaussian_filter_2d (y, v ): pad_width1 = (int ((v.shape[0 ]-1 )/2.0 ), v.shape[0 ]-1 -int ((v.shape[0 ]-1 )/2.0 )) pad_width2 = (int ((v.shape[1 ]-1 )/2.0 ), v.shape[1 ]-1 -int ((v.shape[1 ]-1 )/2.0 )) y_pad = np.pad(y, (pad_width1, pad_width2), mode='edge' ) y_filter = convolve(y_pad, v, mode='valid' ) return y_filter v = get_v_2d(1.0 , 1.0 ) Z_filter = get_gaussian_filter_2d(Z, v) fig, axs = plt.subplots(ncols=1 , nrows=2 , dpi=100 , layout='constrained' , figsize=[5 ,10 ]) axs[0 ].contourf(X, Y, Z, level=50 , cmap='viridis_r' ) axs[0 ].set_xlabel('X' ) axs[0 ].set_ylabel('Y' ) axs[0 ].set_aspect('equal' ) axs[0 ].set_title('Z' ) axs[1 ].contourf(X, Y, Z_filter, level=50 , cmap='viridis_r' ) axs[1 ].set_xlabel('X' ) axs[1 ].set_ylabel('Y' ) axs[1 ].set_aspect('equal' ) axs[0 ].set_title('Z filter' )
下面为代码的运行结果:
参考资料:
https://yixuan.blog/cn/2011/06/convolution/ https://www.zhihu.com/question/22298352 https://blog.csdn.net/qq_36575363/article/details/109684517